Scripting sur OMERO
Les scripts sont accessibles via l’interface web par la petite icône d’engrenage.
OMERO dispose d’une page d’aide sur les scripts: https://omero-guides.readthedocs.io/en/latest/scripts/docs/index.html
Une grande quantité de scripts est disponible. En raison de la quantité élevée de scripts disponibles, de l’absence d’images de test spécifiques, et de l’existence inconstante de documentation spécifique à chaque script (vous devrez quelquefois vous contenter des commentaires présents dans le script), ceux-ci n’ont pas été testés (sauf exceptions spécifiques, mentionnées explicitement) et leur utilisation se fera à vos risques et périls.
La plupart des scripts donnent accès à une petite interface graphique dans laquelle les paramètres sont renseignés.
Certaines équipes apportent une documentation plus détaillée à leurs scripts.
La liste fournie ici ne prétend pas être exhaustive et sera potentiellement amenée à être remise à jour.
Scripts officiels
Consultables ici: https://github.com/ome/omero-scripts/ (branche “develop”, la plus à jour). Il est à noter qu’ils sont régulièrement mis à jour ou corrigés (à rythme variable), et par conséquent, les scripts installés sur notre système ne sont pas systématiquement les dernières mises à jour.
Les scripts décrits ci-dessous ne sont pas tous disponibles dans notre première installation d’OMERO (les rajouter?) (une partie seulement est installée). Les descriptions des scripts sont reprises des commentaires (en anglais).
5 catégories de scripts sont proposées par l’équipe d’OMERO:
Scripts d’analyse (analysis_scripts)
Ces scripts sont dédiés à la production de résultats numériques à partir de fichiers images:
-
“Kymograph.py”: Ce script est destiné à la création de kymographes. Ce script prend en paramètres des images, avec des ROIs linéaires droites (Line) ou en lignes brisées (PolyLine) pour créer des kymographes. Les kymographes sont créées sous la forme de nouvelles images OMERO, monocanal, avec une seule profondeur Z et un seul timestamp T, et de même taille que l’image d’entrée.
-
“Kymograph_Analysis.py”: Ce script est le deuxième script dédié aux kymographes, permettant d’analyser les lignes dessinées sur les images de kymographes qui ont été créées par le script ‘Kymograph.py’.
Note: Pour tous ceux qui se demandent ce qu’est un kymographe en imagerie, la réponse est ici: https://imagej.net/tutorials/generate-and-exploit-kymographs
- “Plot_Profile.py”: Ce script prend en paramètres des images, avec des ROIs linéaires droites (Line) ou en lignes brisées (PolyLine), et permet de sauvegarder l’intensité des canaux sélectionnés dans un fichier CSV.
Scripts d’exportation (export_scripts)
Ces scripts sont dédiés à l’exportation d’images:
-
“Batch_Image_Export.py”: Ce script prend en paramètre les images sélectionnées (ou entrées dans le champ correspondant) et sauvegarde les plans individuels (zoom réglable) dans un fichier zip. Ce script fait l’objet d’un passage dans la section 5 (Exportation d’images, paragraphe 3). S’y reporter pour plus de précisions.
-
“Batch_ROI_Export.py”: Ce script exporte les intensités correspondant aux ROIs pour les images séléctionnées dans un fichier CSV.
-
“Make_Movie.py”: Ce script prend un certain nombre de paramètres et crée un film à partir des images sélectionnées (ou dont l’imageId est entré dans le champ correspondant). Ce film est ensuite placé en pièce jointe sur l’image d’origine. Ce script fait l’objet d’un passage dans la section 5 (Exportation d’images, paragraphe 2). S’y reporter pour plus de précisions.
Scripts dédiés à l’élaboration de figures (figure_scripts)
Ces scripts sont dédiés à l’élaboration de figures, qu’elles soient directement utilisables sur OMERO.figure ou directement sorties en tant que fichiers images:
-
“Movie_Figure.py”: Ce script produit pour une image timelapse une planche contact des différents temps. La planche contact est sauvegardée au format JPG ou PNG (au choix) et attachée en pièce jointe à l’image d’origine.
-
“Movie_ROI_Figure.py”: Ce script prend un certain nombre d’images en paramètre et produit un film des ROIs.
-
“ROI_Split_Figure.py”: Ce script prend un certain nombre d’images en paramètre et affiche les régions en tant qu’images zoomées à côté des images.
-
“Split_View_Figure.py”: Ce script prend un certain nombre d’images et produit une figure des canaux séparés et de l’image mergée, pour chaque image. Une image par rangée. La figure est lisible sur OMERO.figure.
-
“Thumbnail_Figure.py”: Ce script produit une planche contact des vignettes (miniatures) associées aux images sélectionnées (ou dont l’imageId est entré dans le champ correspondant). La planche est sauvegardée en tant que pièce jointe associée à la première image sélectionnée, sous forme de fichier image.
Scripts dédiés à l’importation de métadonnées (import_scripts)
Ces scripts sont utilisés sur les images importées pour l’apport des métadonnées associées aux dites images:
-
“Populate_Metadata.py”: Ce script prend en paramètre un fichier CSV et crée un tableau OMERO (OMERO.table) à partir du dit fichier, avec une ligne par image, par puits (Well), ou par ROI. Une documentation sur ce script est visible ici: https://omero-guides.readthedocs.io/en/latest/upload/docs/metadata-ui.html
-
“Populate_ROI.py”: Ce script prend en paramètre l’identifiant de la plaque (Plate), récupère toutes le mesures associées à la plaque et génère des ROIs à partir de ces mesures. Ce script ne sera utilisable que pour de l’HCS.
Scripts “de niche” (util_scripts)
Ces scripts sont utilisés pour des besoins marginaux, divers et variés:
-
“Channel_Offsets”: Ce script permet d’opérer des décalages dans X, Y, Z, de façon séparée pour chaque canal de l’image ou des images sélectionnées, vraisemblablement à des fins de correction d’alignement. Ce script crée des nouvelles images OMERO, par défaut dans le dataset actuel. Il est possible de les inclure dans un nouveau dataset grâce au champ approprié.
-
“Combine_Images.py”: Ce script prend en paramètre une série d’images (ou stacks) et les associe en une pile d’images afin d’obtenir une image multicanaux, multitemps, tridimensionnelle (plusieurs profondeurs Z). Vous devrez manuellement définir le nombre de canaux, la profondeur et le temps.
-
“Dataset_To_Plate.py”: Ce script permet de convertir un Dataset OMERO en une plaque (Plate), avec une image par puits (Well). Ce script ne servira que pour les pratiquants de la microscopie HCS (High Content Screening).
-
“Images_From_ROIs.py”: Ce script permet d’obtenir des images à partir des ROIs de chaque image sélectionnée. Ce script fait déjà l’objet d’une documentation spécifique (section 8 sur l’utilisation des ROI, paragraphe 2: “Créer des miniatures d’images à partir de ROIs carrées”)
-
“Move_Annotations.py”: Ce script permet de déplacer des annotations appartenant aux images OMERO vers leurs puits (Wells) parents. Ce script ne servira que pour les pratiquants de la microscopie HCS (High Content Screening).
Scripts dédiés à l’annotation (annotation_scripts)
Ces scripts devenus populaires ont été abandonnés par leur équipe originale de développement (les scripts d’origine sont ici: https://github.com/mpievolbio-scicomp/obat). Devant leur utilité, les scripts ci-dessous ont été repris par l’équipe OMERO et regroupés récemment dans cette nouvelle catégorie:
-
“KeyVal_from_csv.py”: Ce script s’applique à un dataset. Il permet d’associer les valeurs d’un fichier CSV attaché au dataset (dans la rubrique “Attachments”) aux images du dataset, sous formes de paires clés-valeurs.
-
“KeyVal_to_csv.py”: Ce script s’applique à un dataset. Il permet de convertir les paires clés/valeurs (“Key-Value Pairs”) des images du dataset (pas du répertoire du dataset) en un fichier de texte brut au format CSV.
-
“Remove_KeyVal.py”: Ce script permet d’enlever tous les dictionnaires des images d’un dataset, et du dataset lui-même.
Ces scripts faisaient déjà l’objet d’une documentation écrite par l’équipe de développement (https://mpievolbio-scicomp.pages.gwdg.de/blog/post/2020-09-03_omerobulkannotation/), toujours d’actualité, le fonctionnement étant identique. Cette même documentation a été reprise pour l’élaboration d’un tutorial interne disponible sur le blog (section 10 sur les métadonnées, passage “Utilisation des scripts de la collection “Omero Bulk Annotation Tools”)
Scripts provenant d’autres sources professionnelles (pour utilisateurs avertis)
Ces scripts proviennent de sources professionnelles (universités et centres de recherche) sans lien avec l’équipe des programmeurs d’OMERO. Normalement utilisables, ils sont toutefois au mieux maintenus de façon plus ou moins régulière, au pire abandonnés par leurs créateurs. Ils disposent généralement (ou pas) de leur propre documentation, au bon gré de leur organisme géniteur. Si vous les utilisez, attendez-vous occasionnellement à ce qu’ils soient inutilisables en raison d’une absence de portage de Python 2.7 vers Python 3 (ou ultérieur). Cela vaudra aussi pour les bibliothèques associées. Il sera préférable de les utiliser avec du recul et des connaissances en programmation et en bibliothèques Python.
Scripts OBAT (OMERO Bulk Annotation Tools)
Dépôt: https://github.com/mpievolbio-scicomp/obat
Ces scripts destinés à l’annotation d’images ont été écrits par le personnel de l’Institut Max Planck de Biologie évolutive (Plön, Allemagne). Malgré leur âge, ils sont encore utilisables. Ils ont déjà été traités à un autre endroit du blog (section 10 sur les métadonnées, passage “Utilisation des scripts de la collection “Omero Bulk Annotation Tools”) ainsi que dans la section précédente et il serait redondant de les repasser en revue ici.
Scripts de Will Moore (développeur d’OME travaillant sur OMERO)
Dépôt: https://github.com/will-moore/python-scripts
Ces scripts ont été écrits par Will Moore (un des développeurs d’OMERO) en réponse à des questions posées sur le forum Image.sc (https://forum.image.sc/). Il y a vraiment de tout. Pas de documentation formalisée, mais les codes contiennent une ligne de commentaire avec le lien du fil du forum concerné.
Scripts du SAERI (South Atlantic Environmental Research Institute)
Dépôt: https://github.com/saeri-ims/omero-docker-compose/tree/master/custom_scripts Documentation des scripts: https://github.com/saeri-ims/omero-docker-compose/blob/master/scripts_documentation.md
Ces scripts sont principalement destinés au tagging des images ainsi que des scripts d’administration destinés à la maintenance des bases de données de tags. Les scripts ne sont toutefois pas formatés pour Python 3 et ultérieur et ne sont pas maintenus. Si vous voulez tester ces scripts, penser à corriger ces erreurs de syntaxe. Commencez par ajouter des parenthèses aux fonctions “print”:
print "item"-> print("item")
Se lancer dans le scripting (réservé aux administrateurs)
Il vous sera possible de coder vous-même vos propres scripts, si vous disposez de connaissances de base en programmation. Plusieurs API sont disponibles (https://omero-guides.readthedocs.io/en/latest/api_usage.html):
- Java
- MATLAB
- Python
- R
Généralement, pour commencer, vous voudrez peut-être examiner certains scripts déjà existants (commencer par les plus simples), apprendre un peu comment ils fonctionnent et essayer de les modifier. Choisissez votre API selon les bibliothèques disponibles et vos besoins.
Test de scripts
Installer des scripts, qu’ils soient récupérés depuis une source externe ou codés “maison” est un privilège d’administrateur. Un script contenant des erreurs fait planter le démarrage du serveur. En conséquence, avant de chercher à vouloir importer un script, il conviendra de se confectionner un environnement de test.
Les scripts OMERO sont installés côté serveur. Vous devrez en conséquence créer votre propre instance serveur-client OMERO. Si vous disposez d’une machine sous Linux, des tutoriaux d’installation d’OMERO.server (https://omero.readthedocs.io/en/stable/sysadmins/unix/server-installation.html) et d’OMERO.web (https://omero.readthedocs.io/en/stable/sysadmins/unix/install-web/web-deployment.html).
La méthode la plus simple est toutefois d’utiliser une installation par conteneurs Docker. Les conteneurs Docker utilisés par notre équipe (à des fins de test) se trouvent ici: https://gitlab.in2p3.fr/fbi-data/dockers-projects
Importer un script (administrateur uniquement)
Il existe 2 méthodes:
-
La plus complexe, en ligne de commande Linux, décrite dans le tuto suivant: https://omero.readthedocs.io/en/stable/developers/scripts/user-guide.html. Cette page dispose en outre de conseils intéressants pour débuter en programmation de scripts.
-
La plus simple, par le biais d’OMERO.insight ou l’interface web, par la fonction “Upload Script”.
-
Pour l’interface web, cette fonction est disponible dans le menu “Scripts” (icône de rouages mécaniques)
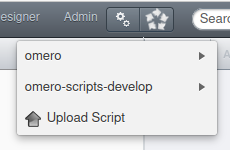
En cliquant sur “Upload Script”, une fenêtre s’affiche, permettant de sélectionner votre script (sur votre disque dur) et le répertoire de destination (sur le serveur)
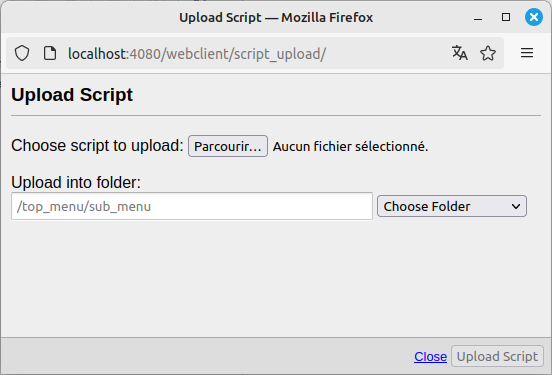
-
Pour OMERO.insight, cliquer sur la petite icône de rouages mécaniques avec une flèche bleue d’upload:

En cliquant sur cette icône, une fenêtre s’affiche, permettant de sélectionner votre script (sur votre disque dur) et le répertoire de destination (sur le serveur)
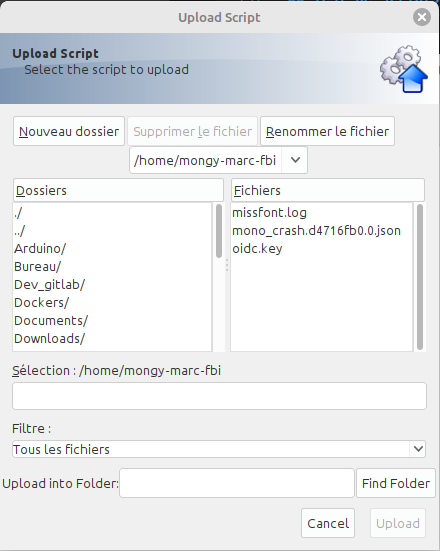
-